Nature | 单细胞测序技术解开肿瘤分型之迷
文章来源:BioArt 作者:赛小记 责任编辑:admin & gaozx 时间:2019-08-10
本文经授权转载自微信公众号BioArt
肿瘤的细胞来源和分型一直是基础研究和临床中的两个难题。了解细胞的起源有助于鉴定致癌基因,根据致癌基因的差异,可以对肿瘤分型,并有助于开发有针对性的药物。
近十年来,得益于基因组学技术,肿瘤的分型经历了从组织学到细胞学到分子学的重要进步。以最为常见的恶性儿童脑瘤,髓母细胞瘤为例。1973年,由于相似的组织特质,髓母细胞瘤最早被归为一类原始神经外胚层肿瘤,但是人们对它的了解极少[1]。
在随后的三十年间,髓母细胞瘤和原始神经外胚层肿瘤之间的差异被逐渐鉴定出来。2007年,WTO将髓母细胞瘤单独列为一类,并通过细胞形态和脑区位置分成了4种亚型。但是这种细胞学上的分型并不能区别肿瘤之间的本质差异[2]。随着基因芯片检测和测序技术的应用,2012年,国际上重新将髓母细胞瘤分为4种分子亚型:WNT型、SHH型、三型和四型[3]。
WNT型和SHH型主要是由于其存在WNT信号通路和SHH信号通路的变异,占30%的髓母细胞瘤病例。这次的分型使得该类型的病人有机会获得靶向药物治疗。但三型和四型的致癌通路却并不明确,且分型也十分困难。
对于三型和四型的特征标记,部分肿瘤能够被鉴定出明显归属一类,另一些肿瘤却难以被分类。WNT和SHH的肿瘤也有较为确定的细胞起源,但是三型和四型的细胞起源却仍是个未知之谜。从目前的存活率来看,WNT亚型患者存活期较长,而三型和四型的存活期却明显偏短。
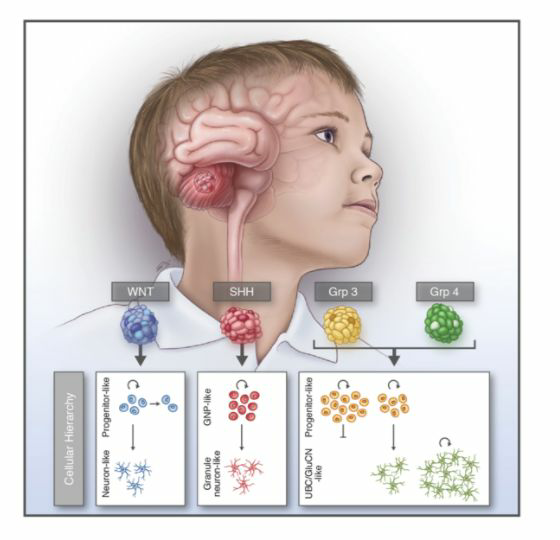
为了探究三型和四型髓母细胞瘤内部的差异,2019年7月25日,美国St.Jude儿童研究医院和麻省总医院的医生和科学家们在Nature杂志上发表文章Resolving medulloblastoma cellular architechture by single-cell genomics,通过单细胞转录组测序技术检测了25个儿童髓母细胞瘤样品。他们发现三型和四型的主要差异在于不同类型的细胞比例,并通过计算推测出三型和四型肿瘤的可能起源和致癌通路。

通过比较三型和四型肿瘤的单细胞图谱,研究者发现三型肿瘤存在未分化和已分化两种细胞类型,而四型肿瘤却只存在是已分化的细胞类型,所以这两种肿瘤的差异其实是不同肿瘤细胞比例的差异,这也解释了为什么部分肿瘤样品会出现介于两者之间的状态。
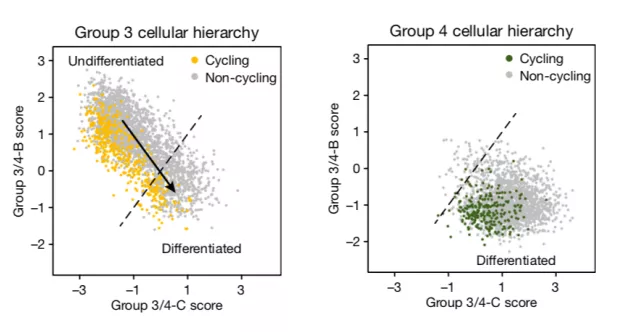
三型肿瘤存在未分化和已分化两种细胞类型,而四型肿瘤却只存在是已分化的细胞类型
同时,通过将肿瘤细胞和发表的成熟脑细胞单细胞图谱进行比较,作者发现SHH型肿瘤和其已知的起源细胞,小脑颗粒神经祖细胞(GNP )最为类似,而四型肿瘤和单极刷细胞(UBC)最为类似,三型肿瘤由于含有大量退分化细胞,没有在成熟的已分化细胞类型中找到它的最佳匹配类型。但其中的分化细胞却更类似 UBC。
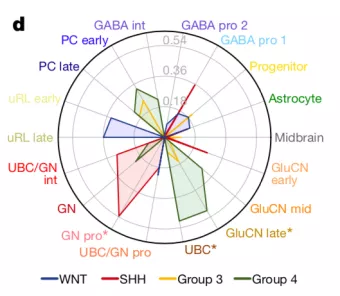
SHH型肿瘤和GNP 最为类似,四型肿瘤和UBC最为类似
最后该研究也鉴定了三型和四型肿瘤的异常表达基因。为将来进一步探究它们的功能打下来基础。
该研究解开了2012年髓母细胞瘤分子分型的遗留问题,当时被视为不同类型的两种肿瘤亚型,现在看来很可能是起源于同一系谱的细胞,具有相似的致癌机理,但发展到不同阶段的肿瘤。这一发现对于未来进一步验证其致癌通路,并提供靶向药物提供了新的思路和基础。
本文的第一作者是来自麻省总医院的 Volker Hovestadt 和 Mariella G. Filbin, 以及美国圣犹大儿童研究医院的 Kyle S. Smith 和 Laure Bihannic。本文的三位通讯作者,Bradley Bernstein, Mario Suva 和 Paul Northcott 都长期致力于运用单细胞和测序技术研究肿瘤学中的各种问题。
来自麻省总医院的 Bradley Bernstein 在2014年首次将单细胞测序技术运用到肿瘤学研究当中,解析了胶质瘤[4],头颈癌[5]和白血病[6]等各种肿瘤细胞的组成, 并提出了各自的成瘤模型。
Mario Suva在Bradley Bernstein实验室完成博士后训练以后,在麻省总医院的独立实验中专注于研究胶质瘤中各种亚型的区别并用单细胞技术再次证明了肿瘤干细胞假说[7-10]。来自圣犹大儿童研究医院的 Paul Northcott 则专注于髓母细胞瘤的研究[11-13]。
原文链接:https://www.nature.com/articles/s41586-019-1434-6
本文经授权转载自微信公众号BioArt
参考文献:
1. Hart, M.N. and K.M. Earle, Primitive neuroectodermal tumors of the brain in children. Cancer, 1973. 32(4): p. 890-7.
2. Kleihues, D.N.L.H.O.O.D.W.W.K.C.P.C.B.A.J.B.W.S.P., 2007 WHO classification of Tumours of the Central Nervous System. 2007.
3. Kool, M., et al., Molecular subgroups of medulloblastoma: an international meta-analysis of transcriptome, genetic aberrations, and clinical data of WNT, SHH, Group 3, and Group 4 medulloblastomas. Acta Neuropathol, 2012. 123(4): p. 473-84.
4. Patel, A.P., et al., Single-cell RNA-seq highlights intratumoral heterogeneity in primary glioblastoma. Science, 2014. 344(6190): p. 1396-401.
5. Puram, S.V., et al., Single-Cell Transcriptomic Analysis of Primary and Metastatic Tumor Ecosystems in Head and Neck Cancer. Cell, 2017. 171(7): p. 1611-1624 e24.
6. van Galen, P., et al., Single-Cell RNA-Seq Reveals AML Hierarchies Relevant to Disease Progression and Immunity. Cell, 2019. 176(6): p. 1265-1281 e24.
7. Tirosh, I., et al., Single-cell RNA-seq supports a developmental hierarchy in human oligodendroglioma. Nature, 2016. 539(7628): p. 309-313.
8. Venteicher, A.S., et al., Decoupling genetics, lineages, and microenvironment in IDH-mutant gliomas by single-cell RNA-seq. Science, 2017. 355(6332).
9. Filbin, M.G., et al., Developmental and oncogenic programs in H3K27M gliomas dissected by single-cell RNA-seq. Science, 2018. 360(6386): p. 331-335.
10. Neftel, C., et al., An Integrative Model of Cellular States, Plasticity, and Genetics for Glioblastoma. Cell, 2019.
11. Northcott, P.A., et al., Subgroup-specific structural variation across 1,000 medulloblastoma genomes. Nature, 2012. 488(7409): p. 49-56.
12. Northcott, P.A., et al., The whole-genome landscape of medulloblastoma subtypes. Nature, 2017. 547(7663): p. 311-317.
13. Northcott, P.A., et al., Enhancer hijacking activates GFI1 family oncogenes in medulloblastoma. Nature, 2014. 511(7510): p. 428-34.
文章来源 | BioArt
撰文 | 赛小记
责编 | 兮
排版 | 良胤
相关文章
浙大儿院血液科:听完讲座开心过小年
浙大儿院小儿血液肿瘤科线上科普后,医护患儿包饺子一起过小年
王景福主任:生殖细胞肿瘤原来有这么多种?
从肿瘤的分化程度来分,生殖细胞肿瘤可以分为两大类:一类是生殖细胞瘤;另一类就是非生殖细胞瘤。
李治中博士:为中国患儿的治疗之路点燃希望
李治中博士首先重点介绍了向日葵儿童的社工项目:在昆明儿童医院建立的首个试点社工站。
我们走过的路(五)
这是向日葵儿童的第一个讲述儿童肿瘤患儿家庭生活的纪实性长篇小说
新疆白血病姑娘与死神的抗争之路
这个世界上只有一种英雄主义:直面生活所给予的一切,并且依然热爱生活!








